셋팅
라이브러리 임포트
데이터 로드
make_blobs()
•
파라미터
◦
n_samples: 표본 데이터의 수
◦
centers: 생성할 클러스터의 수 또는 중심
◦
cluster_std: 클러스터의 표준 편차
•
리턴
◦
X: (n_samples, n_features) 크기의 배열 독립 변수
◦
y: (n_smaples) 크기의 배열 종속 변수
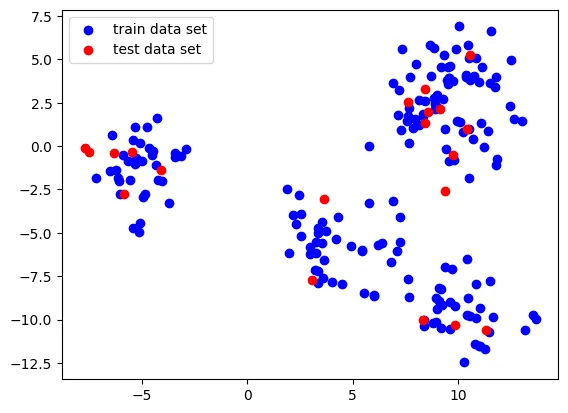
X, _ = make_blobs(n_samples= 200, centers= 5, random_state=4, cluster_std=1.5)
# 훈련 세트와 테스트 세트로 나눕니다
X_train, X_test = train_test_split(X, random_state=5, test_size=.1)
plt.scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
plt.scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
plt.legend()
plt.show()
Python
복사
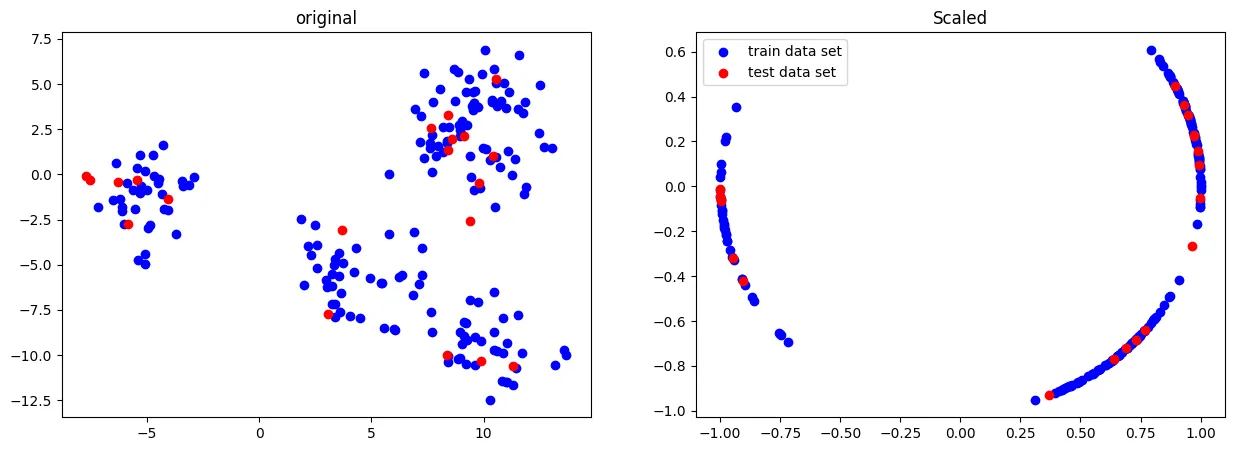
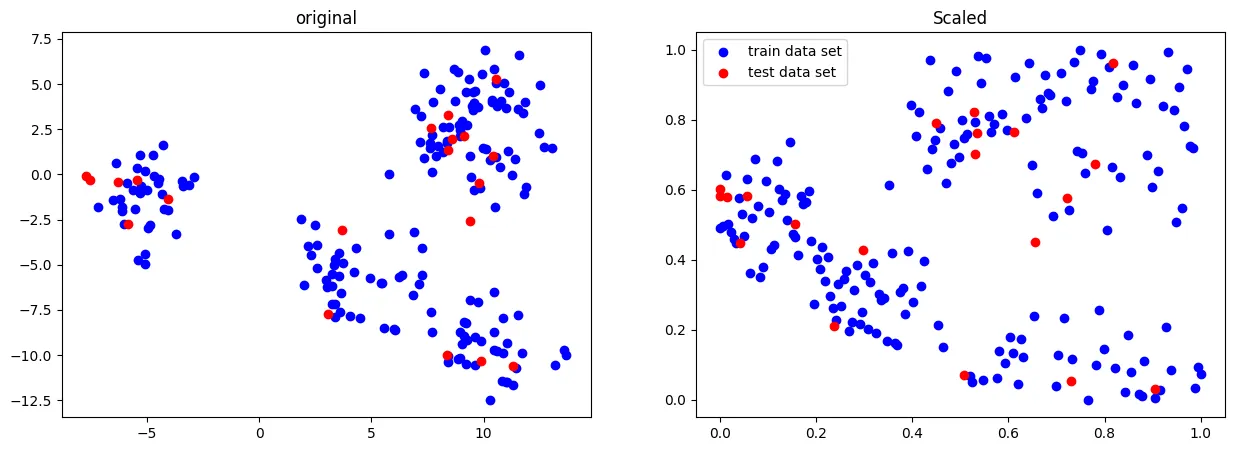
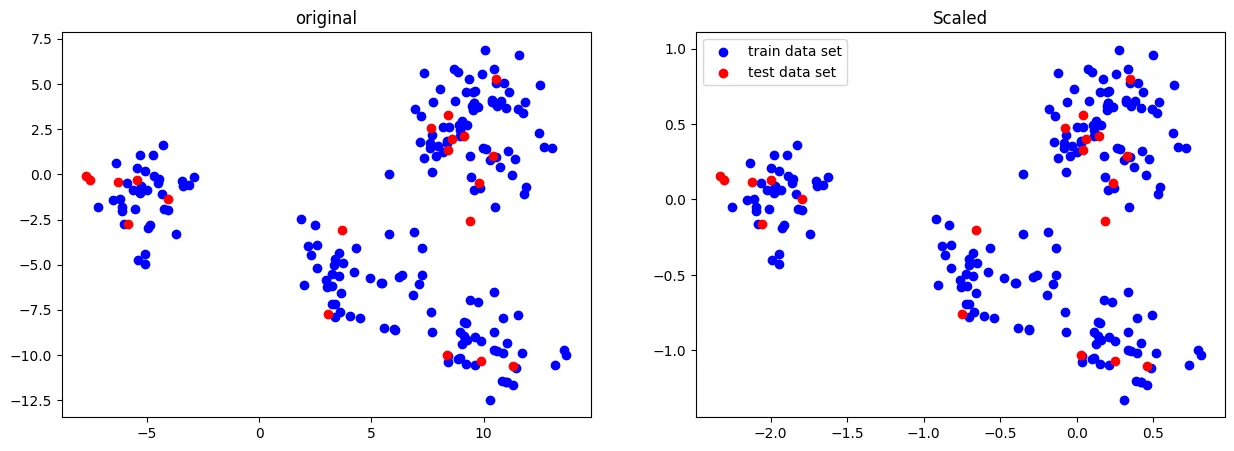
Normalizer(정규화)
•
Normalizer 의 경우 각 샘플(행)마다 적용되는 방식
•
이는 한 행의 모든 특성들 사이의 유클리드 거리(L2 norm)가 1이 되도록 스케일링한다.
•
일반적인 데이터 전처리의 상황에서 사용되는 것이 아니라
•
모델(특히나 딥러닝) 내 학습 벡터에 적용하며,
•
특히나 피쳐들이 다른 단위(키, 나이, 소득 등)라면 더더욱 사용하지 않는다
from sklearn.preprocessing import Normalizer
scaler = Normalizer()
X_scaled = scaler.fit_transform(X_train)
#해당 fit으로 test데이터도 transform 해줍니다
X_test_scaled = scaler.transform(X_test)
fig, ax = plt.subplots(1,2,figsize=[15,5])
# 첫번째 로우 & 왼쪽
ax[0].scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
ax[0].scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
ax[0].set_title('original')
# 첫번째 로우 & 오른쪽
ax[1].scatter(X_scaled[:,0],X_scaled[:,1], c='b', label="train data set")
ax[1].scatter(X_test_scaled[:,0],X_test_scaled[:,1], c='r', label="test data set")
ax[1].set_title('Scaled')
plt.legend()
plt.show()
Python
복사
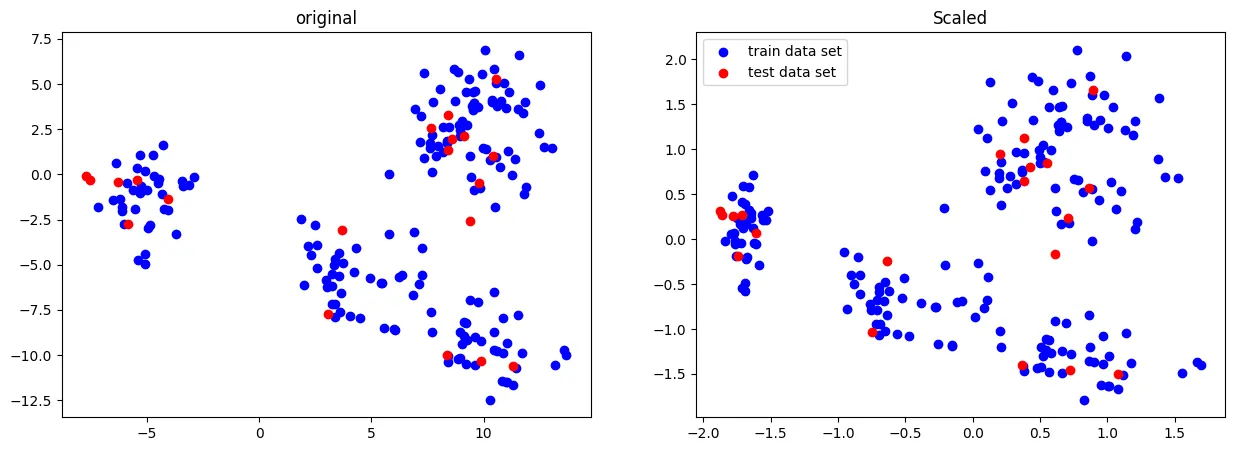
Transformer (변환기)
•
데이터를 정규화(표준화)하는 방법으로, 각 특성(feature)의 평균을 0, 표준편차를 1로 맞추어 변환하는 변환기이다.
•
데이터를 특정한 방식으로 변환하여 머신러닝 모델의 성능을 개선하는 데 사용
정규 분포 (Normal Distribution) : 데이터가 평균값을 중심으로 좌우 대칭적으로 분포하는 형태 - 종 모양
균등 분포 (Uniform Distribution) : 데이터가 특정 구간 내에서 균일하게 분포하는 형태 - 네모 모양
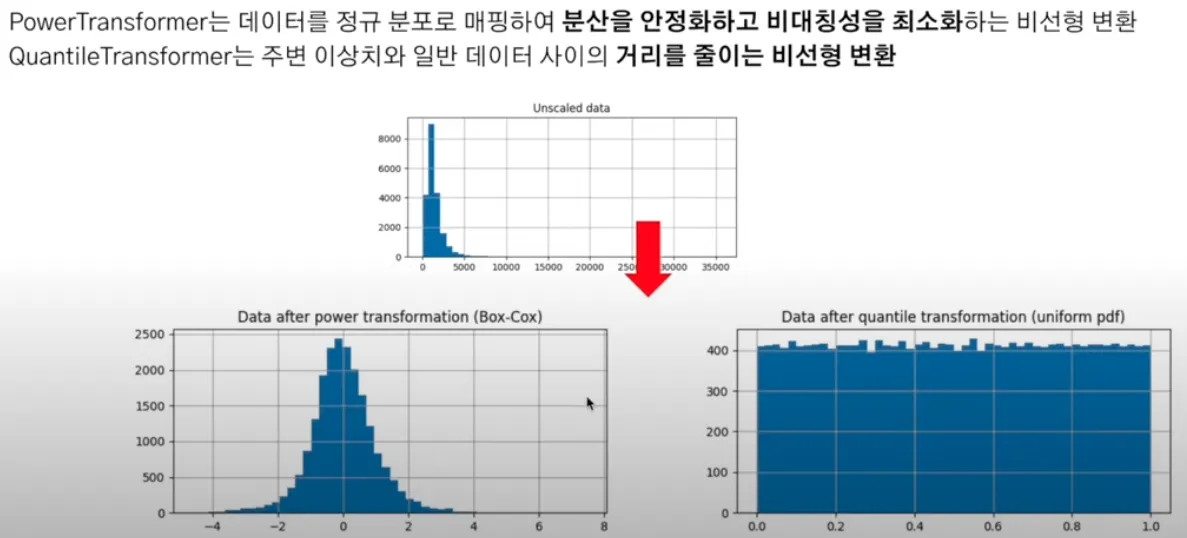
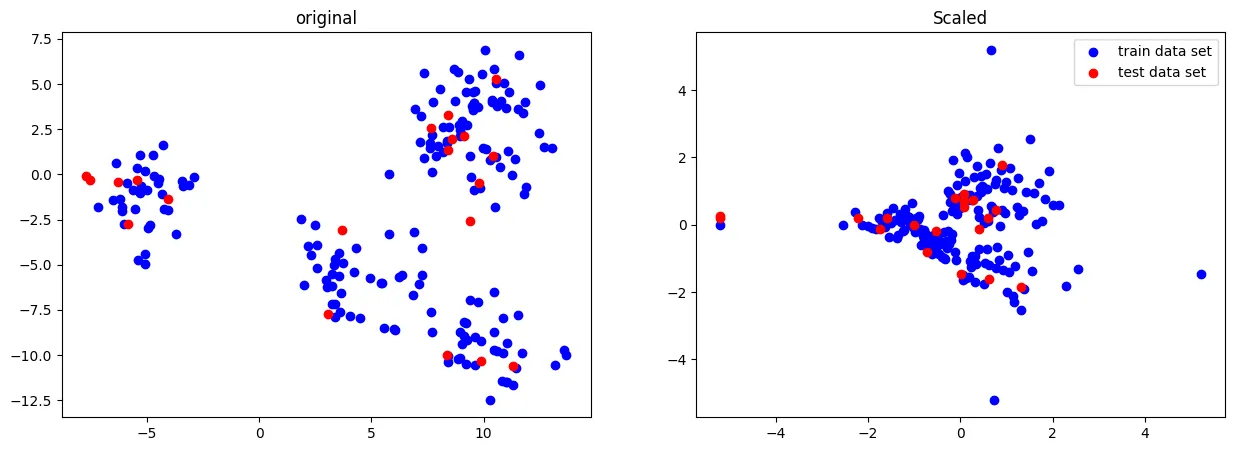
PowerTransformer
•
데이터를 정규 분포에 가깝게 만들기 위해 특정 함수(예: Box-Cox 변환 또는 Yeo-Johnson 변환)를 적용하는 방법
•
사용하는 이유
◦
데이터의 분포가 비대칭적이거나 정규 분포와는 거리가 먼 경우, 모델 성능에 악영향을 미칠 수 있다.
◦
이런 경우 Power Transformer를 사용하여 데이터의 분포를 더 정규에 가깝게 만들 수 있다.
•
데이터의 특성별로 정규분포형태에 가깝도록 변환
from sklearn.preprocessing import PowerTransformer
scaler = PowerTransformer()
# 학습 데이터 스케일링 : 해당 fit으로 test데이터도 transform 해준다.
X_scaled = scaler.fit_transform(X_train)
# fit_transform(X_train)는 X_train 데이터의 분포를 기반으로 스케일링 파라미터를 학습하고, X_train 데이터를 변환하여 X_scaled에 저장
# fit : 학습 데이터의 분포를 분석하여 스케일링에 필요한 파라미터를 계산하고, transform 메서드는 이 파라미터를 사용하여 데이터를 변환
# 테스트 데이터 스케일링
X_test_scaled = scaler.transform(X_test)
# transform(X_test) : X_train에서 학습한 스케일링 파라미터를 사용하여 X_test 데이터를 변환
# 같은 스케일링을 적용하여 모델 학습에서 사용한 데이터와 동일한 스케일로 조정
# 산점도 생성 및 시각화
fig, ax = plt.subplots(1,2,figsize=[15,5])
# 1행 2열의 서브플롯을 생성
# 각 서브플롯의 크기는 15x5 인치
# 첫번째 로우 & 왼쪽
ax[0].scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
ax[0].scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
# X_train[:, 0]과 X_train[:, 1]은 X_train의 첫 두 열을 표시,
# 학습 데이터의 산점도를 파란색('b')으로 표시
# X_test[:, 0]과 X_test[:, 1]은 X_test의 첫 두 열을 표시,
# 테스트 데이터의 산점도를 빨간색('r')으로 표시
ax[0].set_title('original')
# 첫번째 로우 & 오른쪽
ax[1].scatter(X_scaled[:,0],X_scaled[:,1], c='b', label="train data set")
ax[1].scatter(X_test_scaled[:,0],X_test_scaled[:,1], c='r', label="test data set")
# X_scaled[:, 0]과 X_scaled[:, 1]은 스케일링된 학습 데이터의 첫 두 열을 표시,
# 스케일링된 학습 데이터의 산점도를 파란색으로 표시
# X_test_scaled[:, 0]과 X_test_scaled[:, 1]은 스케일링된 테스트 데이터의 첫 두 열을 표시,
# 스케일링된 테스트 데이터의 산점도를 빨간색으로 표시
ax[1].set_title('Scaled')
plt.legend()
plt.show()
Python
복사
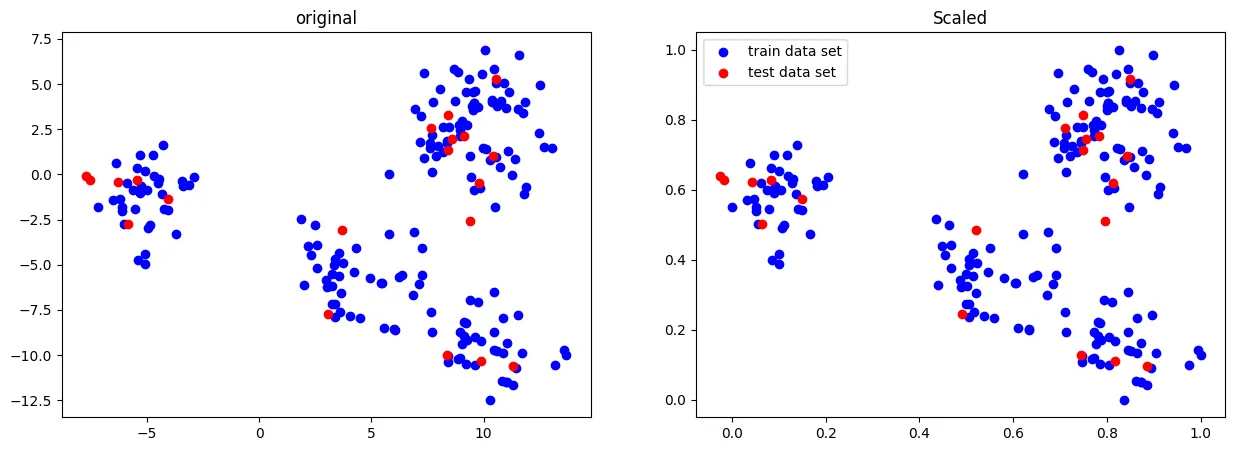
QuantileTransformer
•
데이터를 정렬하고, 특정 분포(예: 정규 분포, 균등 분포)에 따라 데이터의 순위를 매겨 변환하는 방법
•
사용 이유
◦
이상치(outliers)에 민감한 데이터의 경우, 순위 기반 변환을 통해 이상치의 영향을 줄일 수 있다.
◦
데이터를 균등하게 분포시키거나 정규 분포에 맞출 수 있다.
•
작동 방식: 데이터를 정렬한 다음, 각 데이터 포인트를 원하는 분포(정규 분포, 균등 분포 등)에 맞춰 매핑
•
기본적으로 1000개 분위를 사용하여 데이터를 '균등분포' 시킨다.
•
Robust처럼 이상치에 민감하지 않으며, 데이터를 0 ~ 1 사이로 압축한다.
•
기본 사용
from sklearn.preprocessing import QuantileTransformer
scaler = QuantileTransformer()
X_scaled = scaler.fit_transform(X_train)
#해당 fit으로 test데이터도 transform 해줍니다
X_test_scaled = scaler.transform(X_test)
fig, ax = plt.subplots(1,2,figsize=[15,5])
# 첫번째 로우 & 왼쪽
ax[0].scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
ax[0].scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
ax[0].set_title('original')
# 첫번째 로우 & 오른쪽
ax[1].scatter(X_scaled[:,0],X_scaled[:,1], c='b', label="train data set")
ax[1].scatter(X_test_scaled[:,0],X_test_scaled[:,1], c='r', label="test data set")
ax[1].set_title('Scaled')
plt.legend()
plt.show()
/usr/local/lib/python3.9/dist-packages/sklearn/preprocessing/_data.py:2627: UserWarning: n_quantiles (1000) is greater than the total number of samples (180). n_quantiles is set to n_samples.
warnings.warn(
Python
복사
•
정규 분포 형태로 변환
from sklearn.preprocessing import QuantileTransformer
# QuantileTransformer + 정규분포 형태로 변환
scaler = QuantileTransformer(output_distribution = 'normal')
X_scaled = scaler.fit_transform(X_train)
#해당 fit으로 test데이터도 transform 해줍니다
X_test_scaled = scaler.transform(X_test)
fig, ax = plt.subplots(1,2,figsize=[15,5])
# 첫번째 로우 & 왼쪽
ax[0].scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
ax[0].scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
ax[0].set_title('original')
# 첫번째 로우 & 오른쪽
ax[1].scatter(X_scaled[:,0],X_scaled[:,1], c='b', label="train data set")
ax[1].scatter(X_test_scaled[:,0],X_test_scaled[:,1], c='r', label="test data set")
ax[1].set_title('Scaled')
plt.legend()
plt.show()
Python
복사
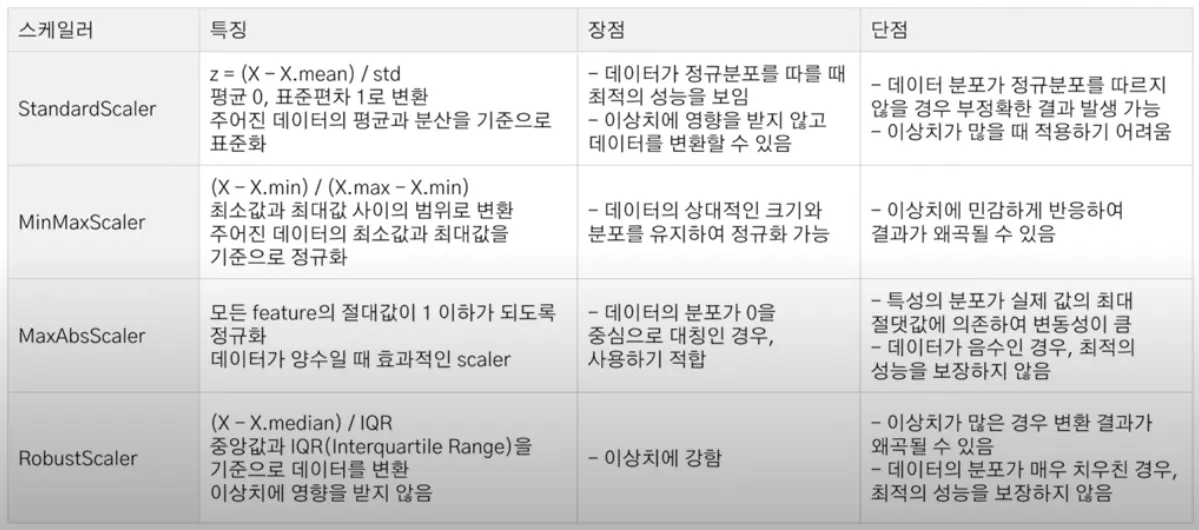
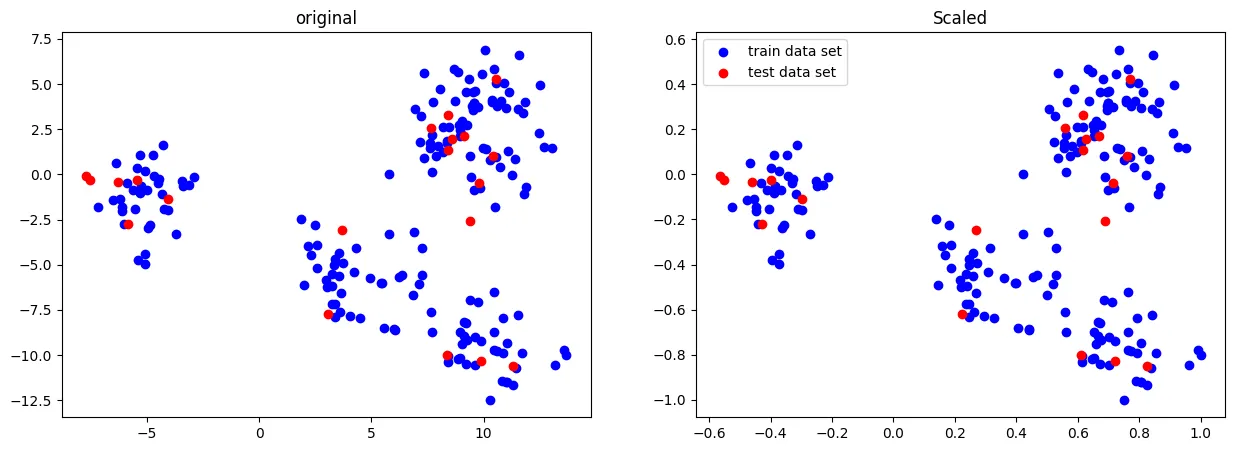
Scaler
•
데이터가 가진 크기와 편차가 다르기 때문에 한 피처의 특징을 너무 많이 반영하거나 패턴을 찾아내는데 문제가 발생한다.
•
따라서 데이터 스케일링을 통해 모든 피처들의 데이터 분포나 범위를 동일하게 조정해줄 필요가 있다.
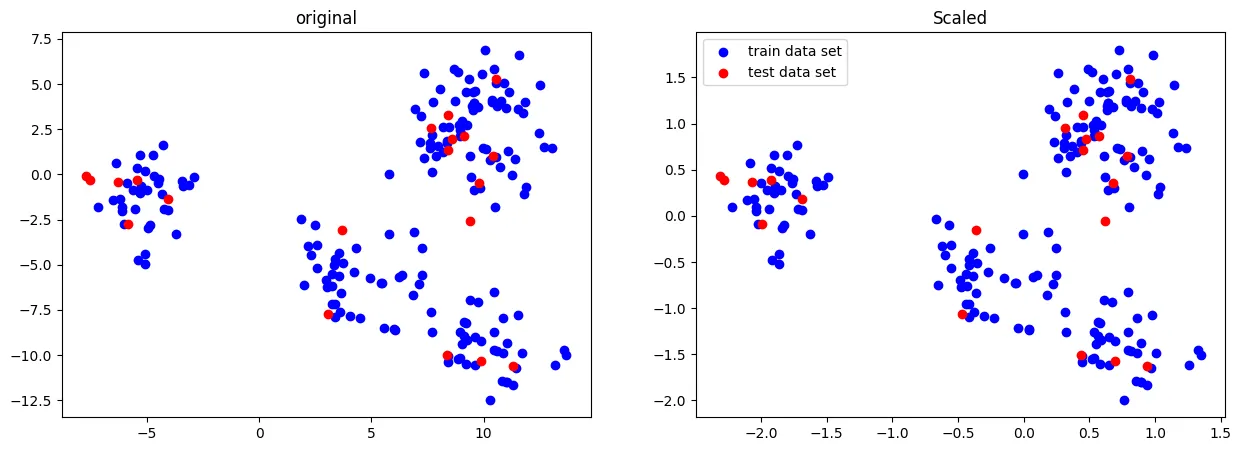
Standard Scaler
•
평균 0, 분산 1로 조정
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X_train)
#해당 fit으로 test데이터도 transform 해줍니다
X_test_scaled = scaler.transform(X_test)
fig, ax = plt.subplots(1,2,figsize=[15,5])
# 첫번째 로우 & 왼쪽
ax[0].scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
ax[0].scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
ax[0].set_title('original')
# 첫번째 로우 & 오른쪽
ax[1].scatter(X_scaled[:,0],X_scaled[:,1], c='b', label="train data set")
ax[1].scatter(X_test_scaled[:,0],X_test_scaled[:,1], c='r', label="test data set")
ax[1].set_title('Scaled')
plt.legend()
plt.show()
Python
복사
Robust Scaler
•
평균과 분산 대신 중간값(median)과 사분위값을 사용
•
중간값과 사분위값을 사용하여 이상치가 있는 데이터에 매우 효과적
from sklearn.preprocessing import RobustScaler
scaler = RobustScaler()
X_scaled = scaler.fit_transform(X_train)
#해당 fit으로 test데이터도 transform 해줍니다
X_test_scaled = scaler.transform(X_test)
fig, ax = plt.subplots(1,2,figsize=[15,5])
# 첫번째 로우 & 왼쪽
ax[0].scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
ax[0].scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
ax[0].set_title('original')
# 첫번째 로우 & 오른쪽
ax[1].scatter(X_scaled[:,0],X_scaled[:,1], c='b', label="train data set")
ax[1].scatter(X_test_scaled[:,0],X_test_scaled[:,1], c='r', label="test data set")
ax[1].set_title('Scaled')
plt.legend()
plt.show()
Python
복사
MinMax Scaler
•
모든 피처들을 0 ~ 1 사이의 데이터 값을 갖도록 만듬
•
음수값이 중요한 데이터일 경우 비권장
•
이상치에 영향을 많이 받는 방법
from sklearn.preprocessing import MinMaxScaler
scaler = MinMaxScaler()
X_scaled = scaler.fit_transform(X_train)
#해당 fit으로 test데이터도 transform 해줍니다
X_test_scaled = scaler.transform(X_test)
fig, ax = plt.subplots(1,2,figsize=[15,5])
# 첫번째 로우 & 왼쪽
ax[0].scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
ax[0].scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
ax[0].set_title('original')
# 첫번째 로우 & 오른쪽
ax[1].scatter(X_scaled[:,0],X_scaled[:,1], c='b', label="train data set")
ax[1].scatter(X_test_scaled[:,0],X_test_scaled[:,1], c='r', label="test data set")
ax[1].set_title('Scaled')
plt.legend()
plt.show()
Python
복사
MaxAbs Scaler
•
모든 피처들의 절댓값이 0과 1 사이에 놓이도록 만들어준다.
•
즉, 0을 기준으로 절댓값이 가장 큰 수가 1 또는 -1의 값을 가지게 된다.
•
이것 역시 이상치에 영향을 많이 받는다.
from sklearn.preprocessing import MaxAbsScaler
scaler = MaxAbsScaler()
X_scaled = scaler.fit_transform(X_train)
#해당 fit으로 test데이터도 transform 해줍니다
X_test_scaled = scaler.transform(X_test)
fig, ax = plt.subplots(1,2,figsize=[15,5])
# 첫번째 로우 & 왼쪽
ax[0].scatter(X_train[:,0],X_train[:,1], c='b', label="train data set")
ax[0].scatter(X_test[:,0],X_test[:,1], c='r', label="test data set")
ax[0].set_title('original')
# 첫번째 로우 & 오른쪽
ax[1].scatter(X_scaled[:,0],X_scaled[:,1], c='b', label="train data set")
ax[1].scatter(X_test_scaled[:,0],X_test_scaled[:,1], c='r', label="test data set")
ax[1].set_title('Scaled')
plt.legend()
plt.show()
Python
복사
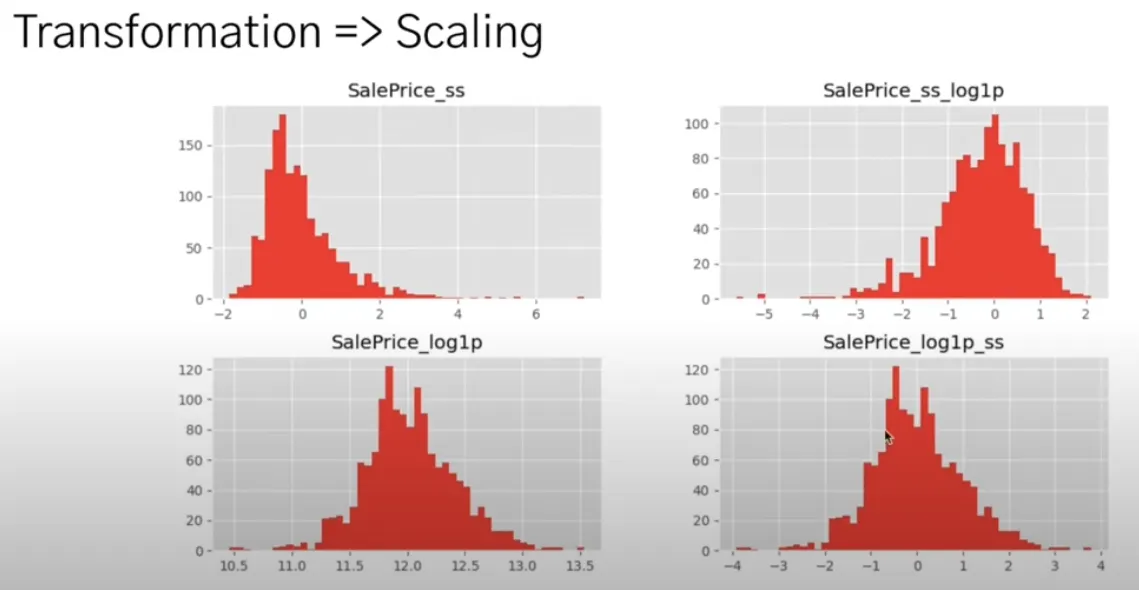
Transformer → Scaler
•
SalePrice_ss -> SalePrice_ss_log1p
◦
Scaler 적용하고, Transformer를 한 경우
◦
두 그래프의 분포도가 달라지는 것을 알 수 있다.(단점)
•
SalePrice_log1p -> SalePrice_log1p_ss
◦
Transformer 적용하고, Scaler를 한 경우
◦
두 그래프의 분포도가 동일한 것을 알 수 있다.(장점)
예제 : 유방암
데이터 로드
from sklearn.datasets import load_breast_cancer
Python
복사
cancer=load_breast_cancer()
dir(cancer)
['DESCR',
'data',
'data_module',
'feature_names',
'filename',
'frame',
'target',
'target_names']
Python
복사
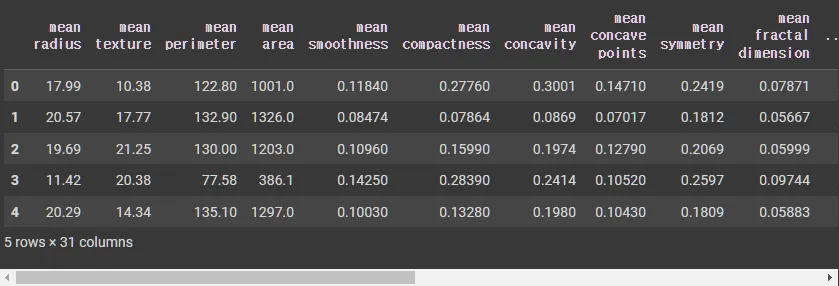
cancer_df = pd.DataFrame(data=cancer.data, columns=cancer.feature_names)
cancer_df['target'] = cancer.target
cancer_df.head()
cancer_df.shape
# (569, 31)
Python
복사
모델 학습
No Scaler
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, test_size=0.2, random_state=3)
dtc = DecisionTreeClassifier()
dtc.fit(X_train, y_train)
no_scaler_score = round(dtc.score(X_test, y_test), 4)
print('모델의 정확도 :', no_scaler_score)
# 모델의 정확도 : 0.9035
X_train.shape
(455, 30)
Python
복사
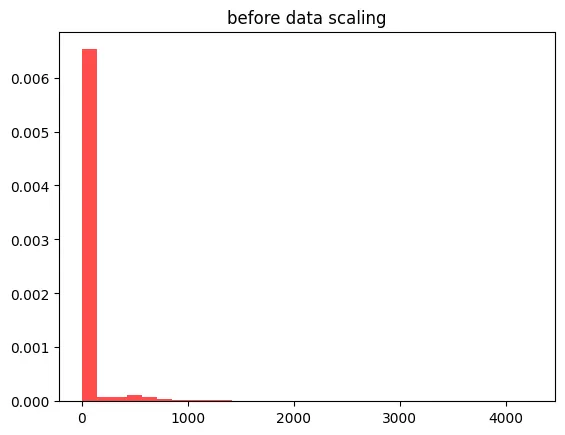
X_train_data = X_train.reshape(13650,1) # (전체 데이터 수[455x30]=1차원(row), 1차원)
plt.hist(X_train_data, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('before data scaling')
plt.show()
Python
복사
Normalizer()
from sklearn.preprocessing import Normalizer
# 스케일링
nor_scaler = Normalizer()
nor_scaler.fit(X_train)
X_train_scaled = nor_scaler.transform(X_train)
X_test_scaled = nor_scaler.transform(X_test)
# 모델 학습
dtc = DecisionTreeClassifier()
dtc.fit(X_train_scaled, y_train)
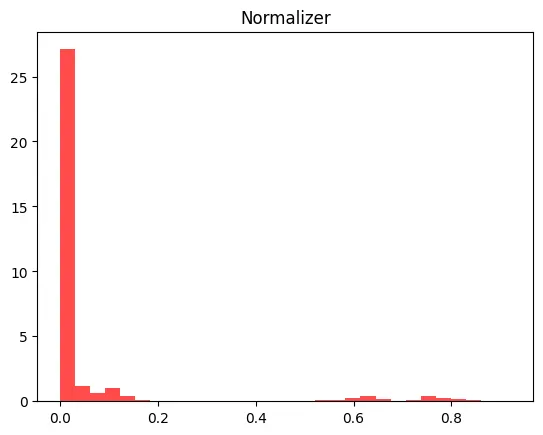
print(f'Normalizer 정확도: {round(dtc.score(X_test_scaled, y_test), 4)} / No Scaler 정확도: {no_scaler_score}')
# Normalizer 정확도: 0.9298 / No Scaler 정확도: 0.9035
Python
복사
X_train_scaled_ss = X_train_scaled.reshape(13650,1)
plt.hist(X_train_scaled_ss, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('Normalizer')
plt.show()
Plain Text
복사
PowerTransformer()
from sklearn.preprocessing import PowerTransformer
# 스케일링
ptf = PowerTransformer()
ptf.fit(X_train)
X_train_scaled = ptf.transform(X_train)
X_test_scaled = ptf.transform(X_test)
# 모델 학습
dtc = DecisionTreeClassifier()
dtc.fit(X_train_scaled, y_train)
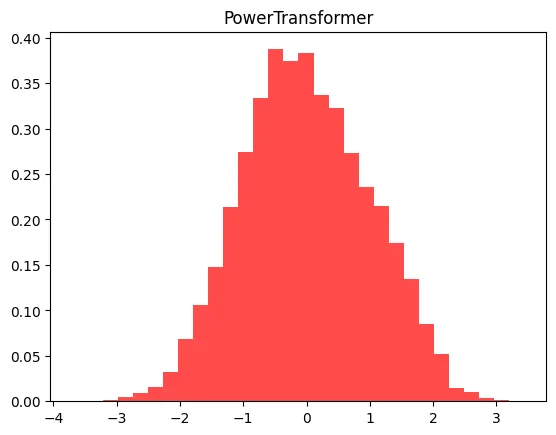
print(f'PowerTransformer 정확도: {round(dtc.score(X_test_scaled, y_test), 4)} / No Scaler 정확도: {no_scaler_score}')
# PowerTransformer 정확도: 0.886 / No Scaler 정확도: 0.9035
Python
복사
X_train_scaled_ss = X_train_scaled.reshape(13650,1)
plt.hist(X_train_scaled_ss, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('PowerTransformer')
plt.show()
Python
복사
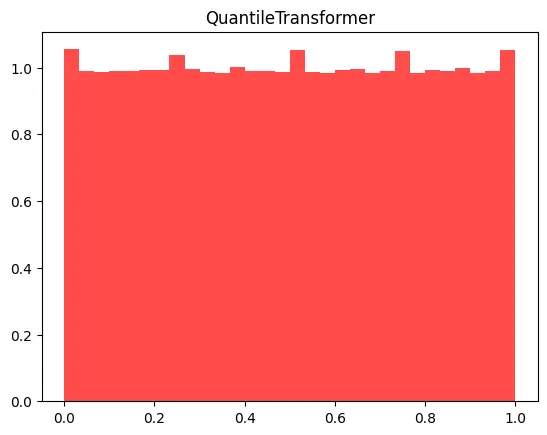
QuantileTransformer()
from sklearn.preprocessing import QuantileTransformer
qtf = QuantileTransformer()
qtf.fit(X_train)
X_train_scaled = qtf.transform(X_train)
X_test_scaled = qtf.transform(X_test)
dtc = DecisionTreeClassifier()
dtc.fit(X_train_scaled, y_train)
print(f'QuantileTransformer 정확도: {round(dtc.score(X_test_scaled, y_test), 4)} / No Scaler 정확도: {no_scaler_score}')
# QuantileTransformer 정확도: 0.8947 / No Scaler 정확도: 0.9035
Python
복사
X_train_scaled_ss = X_train_scaled.reshape(13650,1)
plt.hist(X_train_scaled_ss, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('QuantileTransformer')
plt.show()
Python
복사
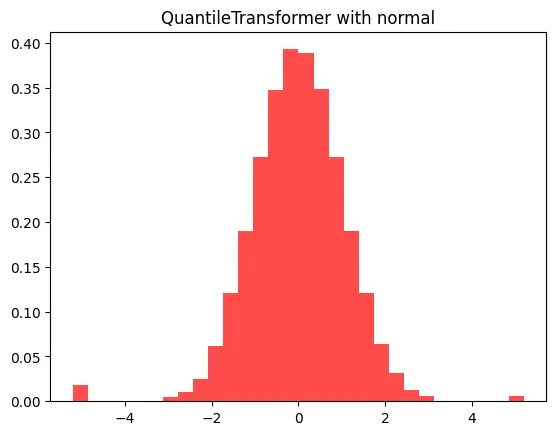
qtf = QuantileTransformer(output_distribution = 'normal')
qtf.fit(X_train)
X_train_scaled = qtf.transform(X_train)
X_test_scaled = qtf.transform(X_test)
dtc = DecisionTreeClassifier()
dtc.fit(X_train_scaled, y_train)
print(f'QuantileTransformer with normal 정확도: {round(dtc.score(X_test_scaled, y_test), 4)} / No Scaler 정확도: {no_scaler_score}')
# QuantileTransformer with normal 정확도: 0.8772 / No Scaler 정확도: 0.9035
Python
복사
X_train_scaled_ss = X_train_scaled.reshape(13650,1)
plt.hist(X_train_scaled_ss, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('QuantileTransformer with normal')
plt.show()
Python
복사
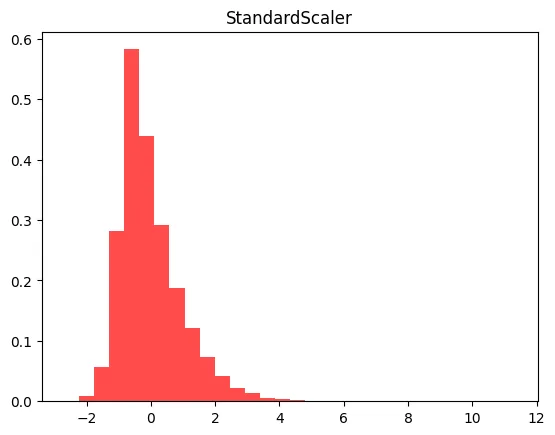
StandardScaler()
from sklearn.preprocessing import StandardScaler
std = StandardScaler()
std.fit(X_train)
X_train_scaled = std.transform(X_train)
X_test_scaled = std.transform(X_test)
dtc = DecisionTreeClassifier()
dtc.fit(X_train_scaled, y_train)
print(f'StandardScaler 정확도: {round(dtc.score(X_test_scaled, y_test), 4)} / No Scaler 정확도: {no_scaler_score}')
# StandardScaler 정확도: 0.9123 / No Scaler 정확도: 0.9035
Python
복사
X_train_scaled_ss = X_train_scaled.reshape(13650,1)
plt.hist(X_train_scaled_ss, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('StandardScaler')
plt.show()
Python
복사
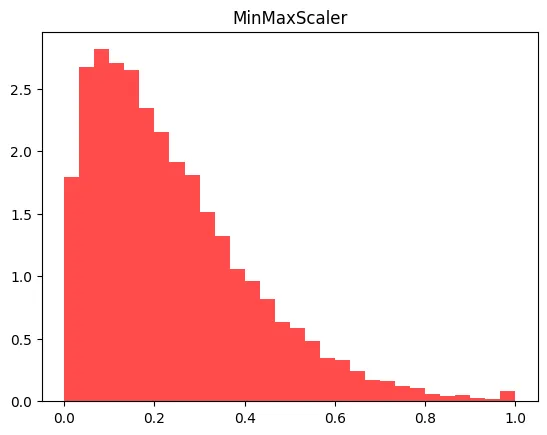
MinMaxScaler()
from sklearn.preprocessing import MinMaxScaler
mms = MinMaxScaler()
mms.fit(X_train)
X_train_scaled = mms.transform(X_train)
X_test_scaled = mms.transform(X_test)
dtc = DecisionTreeClassifier()
dtc.fit(X_train_scaled, y_train)
print(f'MinMaxScaler 정확도: {round(dtc.score(X_test_scaled, y_test), 4)} / No Scaler 정확도: {no_scaler_score}')
# MinMaxScaler 정확도: 0.886 / No Scaler 정확도: 0.9035
Python
복사
X_train_scaled_ss = X_train_scaled.reshape(13650,1)
plt.hist(X_train_scaled_ss, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('MinMaxScaler')
plt.show()
Python
복사
RobustScaler()
from sklearn.preprocessing import RobustScaler
rbs = RobustScaler()
X_train_scaled = rbs.fit_transform(X_train)
X_test_scaled = rbs.transform(X_test)
dtc = DecisionTreeClassifier()
dtc.fit(X_train_scaled, y_train)
print(f'RobustScaler 정확도: {round(dtc.score(X_test_scaled, y_test), 4)} / No Scaler 정확도: {no_scaler_score}')
# RobustScaler 정확도: 0.9035 / No Scaler 정확도: 0.9035
Python
복사
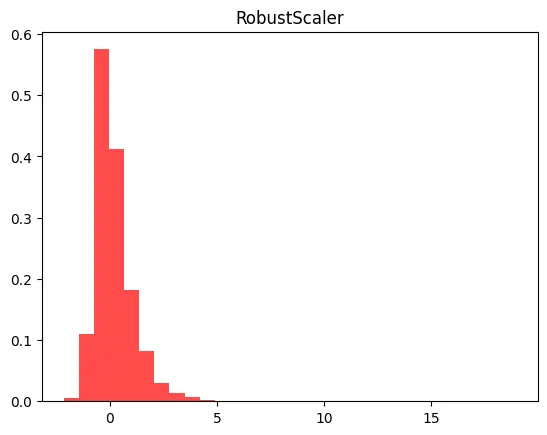
X_train_scaled_ss = X_train_scaled.reshape(13650,1)
plt.hist(X_train_scaled_ss, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('RobustScaler')
plt.show()
Python
복사
MaxAbsScaler()
from sklearn.preprocessing import MaxAbsScaler
mas = MaxAbsScaler()
mas.fit(X_train)
X_train_scaled = mas.transform(X_train)
X_test_scaled = mas.transform(X_test)
dtc = DecisionTreeClassifier()
dtc.fit(X_train_scaled, y_train)
print(f'RobustScaler 정확도: {round(dtc.score(X_test_scaled, y_test), 4)} / No Scaler 정확도: {no_scaler_score}')
# RobustScaler 정확도: 0.8947 / No Scaler 정확도: 0.9035
Python
복사
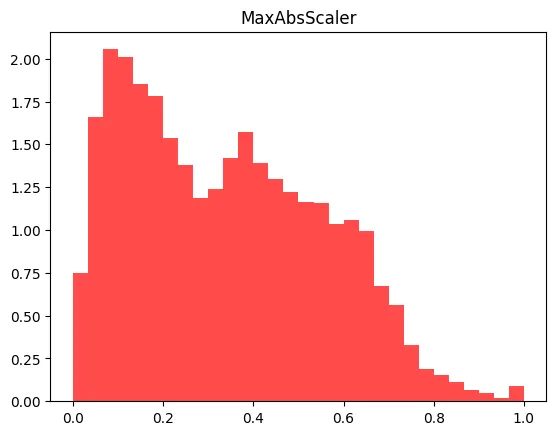
X_train_scaled_ss = X_train_scaled.reshape(13650,1)
plt.hist(X_train_scaled_ss, bins=30, color= 'red', alpha = 0.7, density = True)
plt.title('MaxAbsScaler')
plt.show()
Python
복사